Resumo
A disseminação de doenças infecciosas e resistência antimicrobiana representa uma ameaça significativa, especialmente em países em desenvolvimento onde as técnicas tradicionais de impressão digital de DNA geralmente não estão disponíveis. Propomos uma nova abordagem usando algoritmos de programação dinâmica para comparar sequências de suscetibilidade antimicrobiana e identificar grupos de micróbios relacionados.
Teste antimicrobiano e geração de string ATB para patógenos selecionados. Teste antimicrobiano e geração de string ATB para patógenos selecionados.
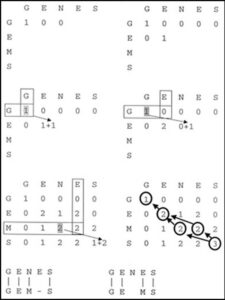
Comparação de sequências usando programação dinâmica. Comparação de sequências usando programação dinâmica.

Cálculo da distância mínima de edição para comparação de sequências. Cálculo da distância mínima de edição para comparação de sequências.

Agrupamento de cepas de Staphylococcus aureus de 29 casos de pneumonia nosocomial. Agrupamento de cepas de Staphylococcus aureus de 29 casos de pneumonia nosocomial.

Identificação de um grande grupo com 16 cepas idênticas de Staphylococcus aureus de 29 casos de pneumonia nosocomial.
Identificação de um grande grupo com 16 cepas idênticas de Staphylococcus aureus de 29 casos de pneumonia nosocomial.

Agrupamento de cepas de Klebsiella pneumoniae de 21 casos de infecção da corrente sanguínea.
Agrupamento de cepas de Klebsiella pneumoniae de 21 casos de infecção da corrente sanguínea.
Todos os autores : Nenhuma divulgação relatada


Comments are closed.